
R 개발자가 ‘R-universe’로 패키지 생태계 운영하는 법
R 개발자가 ‘R-universe’로 패키지 생태계 운영하는 법 관련




R 패키지는 R에서 사용할 수 있는 기능들의 모음으로, 다른 프로그래밍 언어의 라이브러리와 유사한 개념이다. R은 일반적인 프로그래밍 언어와는 살짝 결이 다르고, 매트랩(Matlab), SASS와 같이 통계 분석의 목적을 가지는 프로그램에 가깝다. 그러나 다양한 도메인에서 데이터 분석에 필요한 확장성을 가졌고, ‘base’라 부르는 기본 제공 기능 외에도 여러 기능이 추가됐다.
이번 글에서는 R-universe 플랫폼에서 배포하는 패키지를 예시로, R 개발자가 어떻게 생태계를 운영하고 있는지 살펴보고자 한다.
R 패키지
현재 약 10만 개가 넘는 R 패키지가 있지만, 그래도 한 번쯤은 들어봤을 법한 패키지를 예시로 소개하고자 한다. 먼저 ‘ggplot2’라는 패키지를 사용해, 산점도를 그리는 예시 코드와 그 결과다.
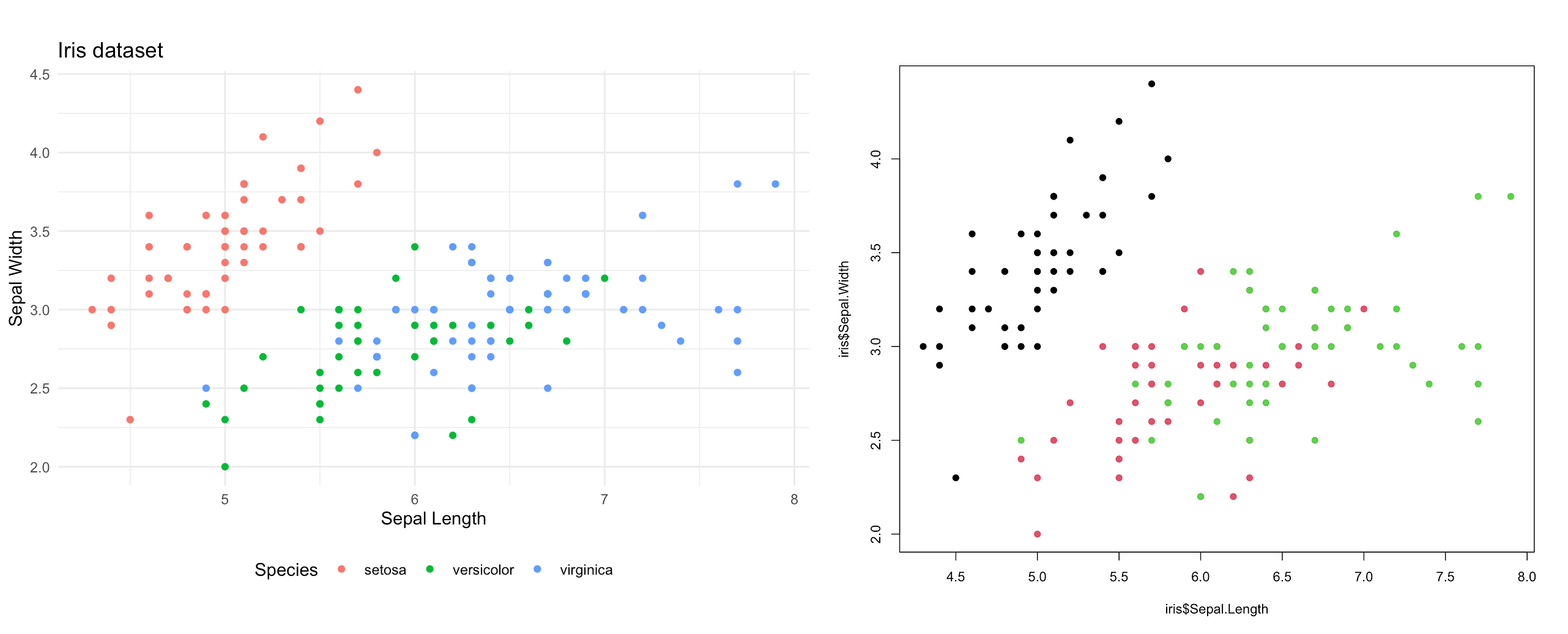
결과에서 아래 오른쪽 그림은 ‘ggplot’을 사용하지 않고 기본 기능만으로 그린 그림이다. 의미 전달은 큰 차이가 없지만 ‘ggplot’ 패키지를 사용하면 코드를 작성하는 편의성은 물론, 시각화에서 더욱 다양한 요소를 활용할 수 있다.
R 패키지 만들기
특히 R을 사용하다 보면 본인의 도메인에서만 쓰는 통계 분석이나, 데이터 시각화용 코드, 혹은 데이터베이스, API를 활용하기 위한 코드를 맨 처음부터 만들어야 하는 경우가 있다. 이를 다른 사람들도 함께 쓸 수 있게 공유하면, 직접 R 패키지를 만드는 경지에 도달하기도 한다.
이렇게 만든 R 패키지를 다른 사람들이 설치, 사용할 수 있도록 온라인에 공개하는 방법은 4가지가 있다.
- (CRAN 같은) Mirror(여기서 Mirror는 다른 페이지의 내용을 다른 URL로 호스팅 하는 미러 사이트로, 이후 “미러”라고 표시)
- (깃허브 같은) VCS(Version Control System)
- 압축 파일
- 텍스트
이 중 3, 4번은 제한된 사용자와 공유하는 경우로, 3번 압축파일은 대학교에서 강의 혹은 시험을 위해 커스텀하게 만든 함수만 사용하게 하는 사례가 있다. 4번 텍스트는 금융이나 의료 기관처럼 데이터 보안이 중요해 분리망 같은 규정과 제한이 있는 곳이다. 보통 .txt 형식의 파일만 내부로 반입 가능한 사례가 있다. 즉, 압축 파일이나 텍스트는 특수한 경우로, R 패키지 중 99%는 미러나 VCS를 통해 공유된다.
VCS를 사용해 R 패키지를 공유하는 방법은 상대적으로 쉽다. 내가 사용하는 깃허브(Github)를 기준으로, 개인이 리포지토리를 만들고 작업한 R 패키지를 커밋하여 올리면 끝이다. 말 그대로 R 패키지의 형식만 지킨다면 누구나 제한 없이 만들 수 있다. ‘README’를 제외한 패키지에 필요한 최소한의 파일만 올리는 것도 가능하다.
두 번째로 R 패키지를 호스팅하고 배포하는 플랫폼 ‘미러’는 공동으로 관리되는 저장소다. 보통은 비영리, 연구 단체 등에 의해 운영된다. 이 미러는 패키지의 목적과 성향에 따라 조금씩 나뉘는데, R 패키지의 다수를 차지하는 CRAN(The comprehensive R Archive Network), 생명정보학 분야에 특화된 Bioconductor, 혹은 오래된 통계 패키지들을 담고 있는 R-forge 등이 있다.
대신 미러는 패키지를 게시하기 위해 제시하는 기준을 만족해야 한다. 패키지에 대한 문서화가 잘 되어 있는지, 오류를 방지하기 위한 테스트가 작성되어 있는지, 다양한 OS에서 문제없이 빌드가 가능한지 등 R 패키지의 품질을 증명해야 등재될 수 있다. 이는 공식 인증을 받는 것이라 꽤 까다로운 프로세스를 거친다. 그렇다고 깃허브에 올라온 R 패키지가 좋지 않다는 건 아니고, 학술 논문으로 치면 Arxiv(=깃허브)와 일반 SCI 저널(=미러)의 차이 정도다.
또한 R 개발자 사이에서 ‘CRAN 기여자’는 꽤 많은 의미를 갖고 있어서, 상대적으로 패키지를 올리기 쉬운 파이썬(Python)의 PyPI, 자바스크립트(Javascript)의 NPM 등 다른 프로그래밍 언어와 R이 차별되는 부분 중 하나다.
CRAN과 깃허브
R 패키지를 CRAN에 올리는 것은 tidyverse 같은 분석용 R 패키지의 코드를 다루는 것 외에도, devtools나 usethis와 같은 개발용 R 패키지를 활용해야 한다. 그만큼 다른 성향의 경험이 필요하고, CRAN에 게시하기까지의 프로세스가 까다롭다. 그래서 많은 R 개발자들은 제한이 없는 깃허브에 올리는 것을 선호한다.
문제는 이렇게 깃허브에 R 패키지를 올리면 (나의 경우를 포함해) 개발자가 직접 꾸준히 관리하지 않는 이상 방치되는 경우가 많다. 사용자는 이러한 역할을 하는 패키지가 있다는 것조차 몰라 잊혀지는 일이 생긴다. 이에 R 패키지 정보를 확인하는 ‘rdrr.io’라는 검색 페이지가 있지만, 패키지를 단위로 큐레이션 하다 보니 파편화되어 있는 R 패키지와 함수 매뉴얼 이상의 정보를 얻을 수 없어 아쉬움이 있다.
최근 ‘useR!2024’라는 컨퍼런스에 다녀왔는데, 토픽 중 하나가 R-universe였다. 이는 tidyverse와 같은 R 패키지 생태계로부터 영감을 받은 것으로, “개인이 만든 패키지들을 하나의 universe로 구성하자”라는 목적이 있다. 좀 더 쉽게 설명하기 위해 예시를 들어보자.
평소 나에게 많은 영감을 주는 R 개발자 ‘Dean Attali’의 R-universe 첫 화면은 아래와 같이 구성되어 있다.
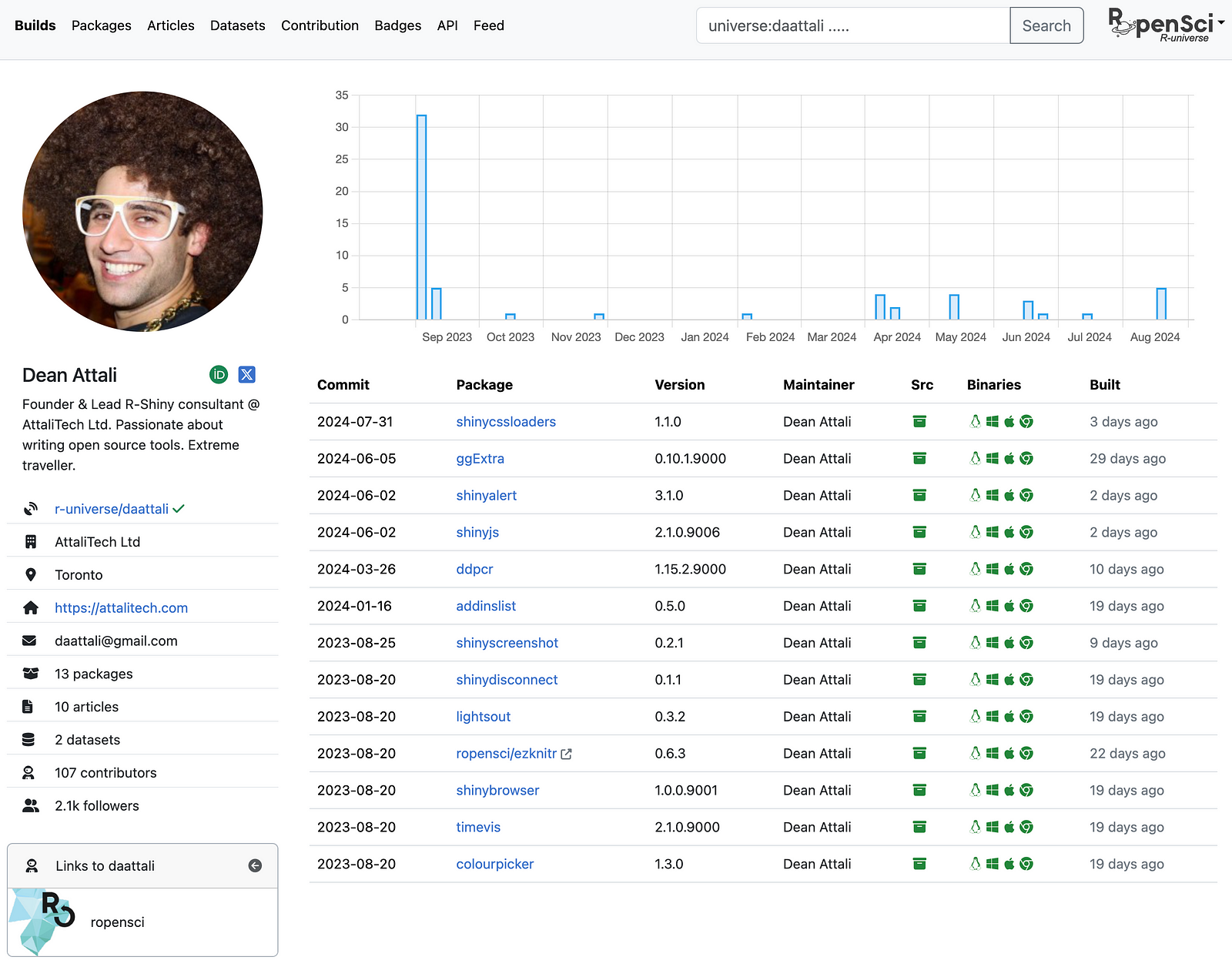
이는 이 사람이 어떤 R 패키지를 만들었는지 알려주고, 각 패키지의 소스 코드와 리눅스, 윈도우, 맥 OS에서 사용 가능한 바이너리 형태의 패키지를 제공하는 퍼스널 미러 역할을 한다. 동시에 공유한 데이터셋과 작성한 R 패키지 관련 아티클 등 R 관련 작업 정보를 한 번에 볼 수 있다.
무엇보다 R-universe의 가장 큰 장점은 CRAN 같은 미러가 아닌, 깃허브 같은 VCS의 R 패키지들로도 개인의 universe를 구성할 수 있다는 점이다. 그렇다면 어떻게 깃허브에 올린 R 패키지를 퍼스널 R-universe에 포함시킬 수 있을까? 단계별로 살펴보자.
퍼스널 ‘R-universe’ 만들기
사실 퍼스널 R-universe는 만약 이전에 R 패키지를 CRAN에 올린 적이 있다면, 이 정보를 바탕으로 R-universe에서 자동으로 생성해 준다. 그러나 자동으로 만들어진 내용은 CRAN 혹은 Bioconductor에 올린 R 패키지만 해당된다. 다른 미러나 깃허브에 R 패키지를 만들었다면 이어지는 내용을 따라 추가할 수 있다.
1) 깃허브 리포지토리 만들기
제일 먼저 해야 하는 일은 깃허브 리포지토리를 만드는 것이다. R-universe에 개인 페이지를 생성하기 위해선 개인 리포지토리를 만들어야 한다. 퍼스널 R-universe를 생성하는 과정은 다음과 같다.
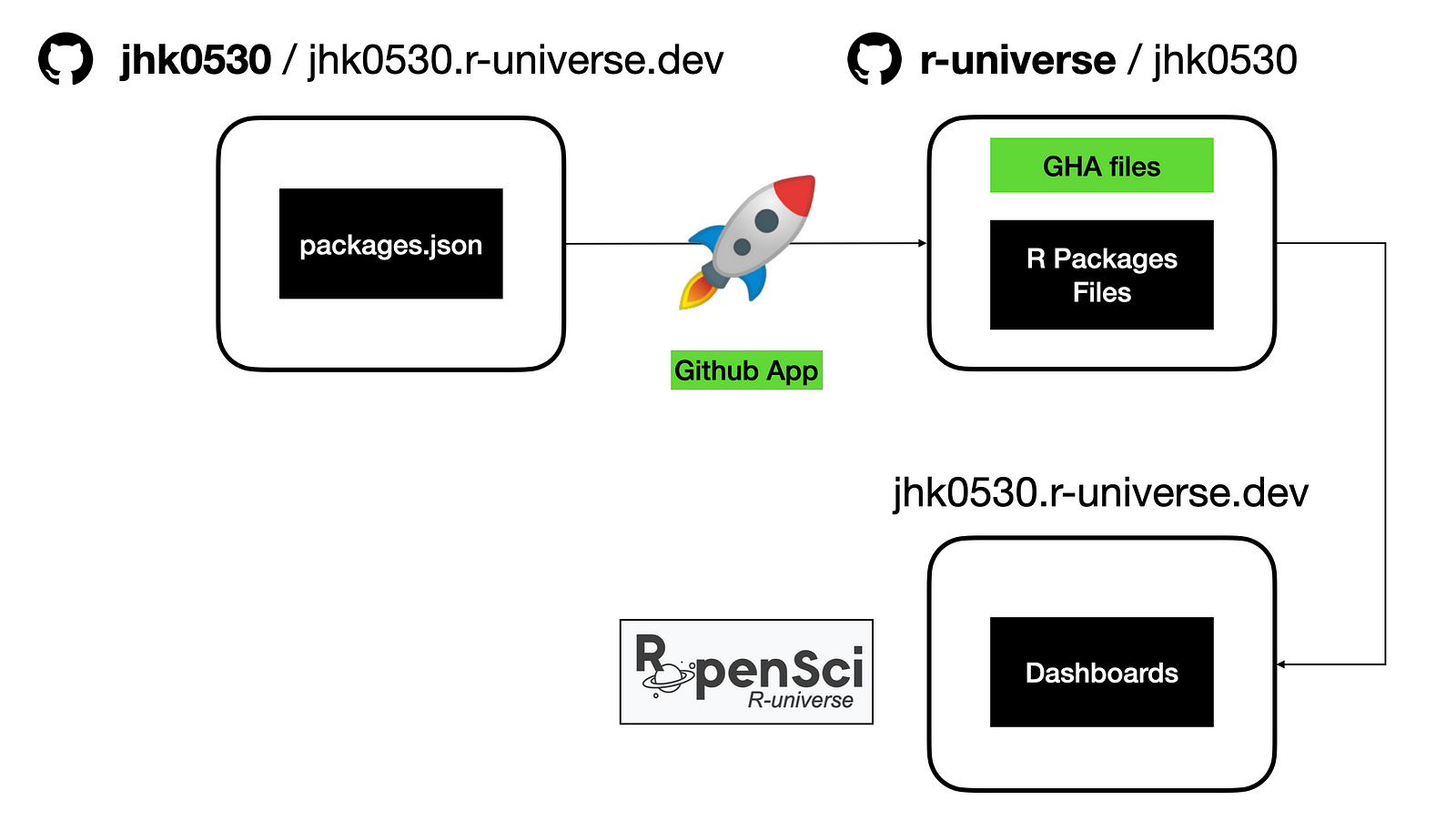
예를 들어, R-universe 인프라팀이 만든 깃허브 앱(Github App)은 내 개인 리포지토리인 ‘jhk0530.r-universe.dev’ 리포지토리에 설치되어, packages.json이라는 파일의 내용을 인식한다. 이 안에는 universe에 추가할 패키지별 이름과 깃허브 리포지토리 주소를 포함해야 한다.
각 패키지의 깃허브 리포지토리에는 README, DESCRIPTION 등 R 패키지를 설명하는 메타 데이터를 포함하고 있어, 깃허브 앱은 이를 활용해 R-universe의 jhk0530이라는 저장소에 내용을 추가 및 업데이트한다.
마지막으로 이 데이터들은 R-universe의 jhk0530이라는 저장소에서 GHA(Github Action)를 사용하여, R-opensci 재단에서 제공, 운영하는 R-universe 웹 페이지에 반영된다.
2) packages.json 만들기
packages.json은 레지스트리 파일로 패키지 이름과 URL을 구조화하여 담고 있다. 이름과 URL 외에도 여러 추가 옵션이 가능하지만, 깃허브에서 브랜치를 사용해 개발하는 것이 아니라면 신경 쓰지 않아도 좋다.내 packages.json의 내용은 아래와 같다. 각각 polaroid와 shinyCyJS는 CRAN에 올린 R 패키지, shinyReadme와 gemini.R은 깃허브에 올린 R 패키지다. 이 내용을 작성하기 전에는 polaroid와 shinyCyJS 의 정보만 내 R-universe 페이지에 있었다.
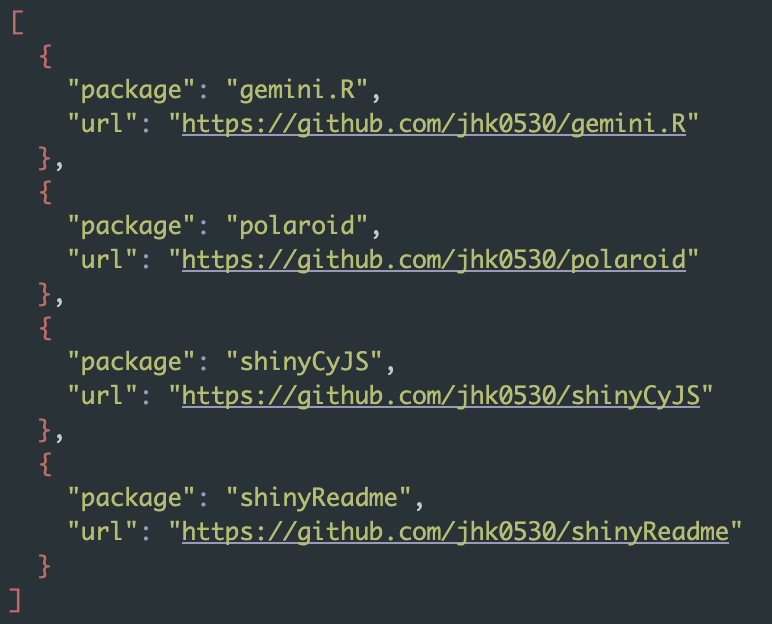
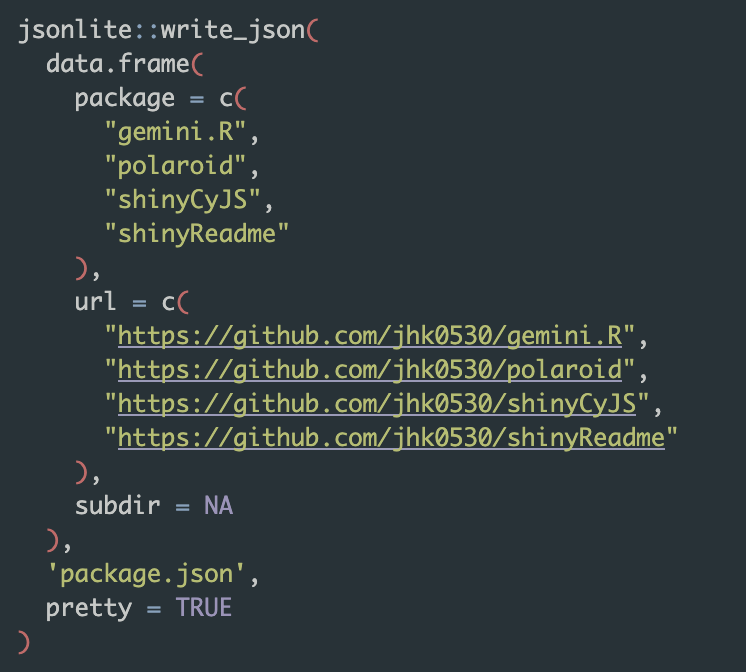
json 데이터를 다뤄본 사람이라면 낯설지 않겠지만, tidyverse 같은 일반적인 R을 다루는 사람에게는 json 파일이 낯설 수 있다. 그래서 R-universe의 공식 매뉴얼에서는 jsonlite 패키지의 write_json 함수 사용을 권장한다. 이어지는 코드는 위 packages.json 파일을 만드는 데 사용한 R 코드다.
3) 깃허브 앱 설치하기
R-universe 인프라팀이 만든 깃허브 앱은 링크 (apps/r-universe)에서 ‘내가 소유 권한이 있는’ 깃허브 계정에 설치할 수 있다. 지금은 내 개인 계정에 universe를 만들기 위한 목적이므로, jhk0530에만 추가한다.
이후 앱이 어떤 리포지토리에 접근할 수 있게 할지 권한을 설정할 수 있는데, 아래 이미지처럼 하나씩 설정하지 말고 ‘All’로 설정하는 것이 좋다. 이 앱이 필요로 하는 권한은 리포지토리의 커밋 기록을 읽고 쓰는 것(read and write commit statuses)밖에 없으니 All로 해도 문제가 없다.
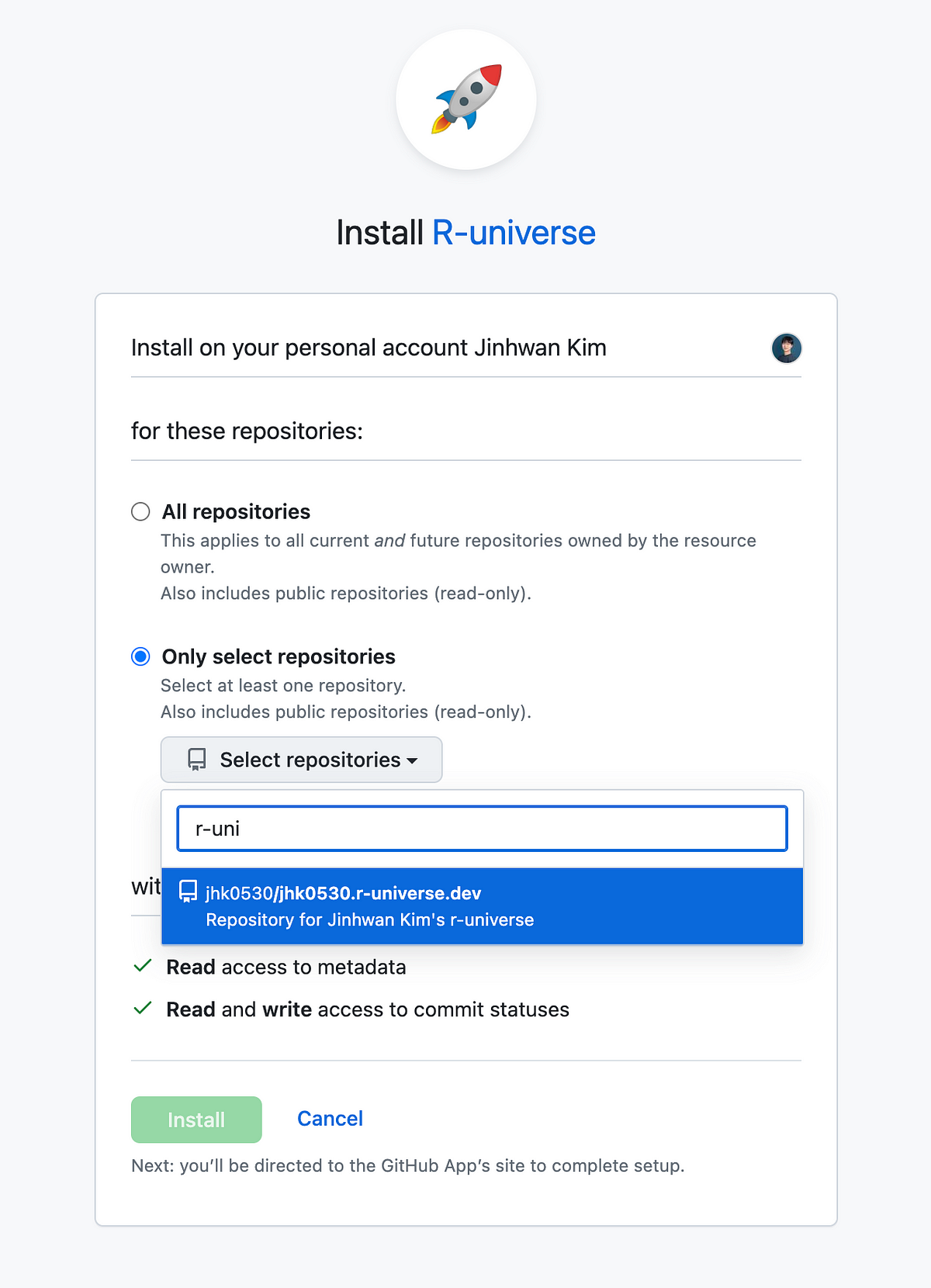
혹시나 Select에서 All로 (혹은 반대로) 바꾸려면, 오른쪽 이미지처럼 Github settings > Integrations > Applications를 통해 설정할 수 있다.
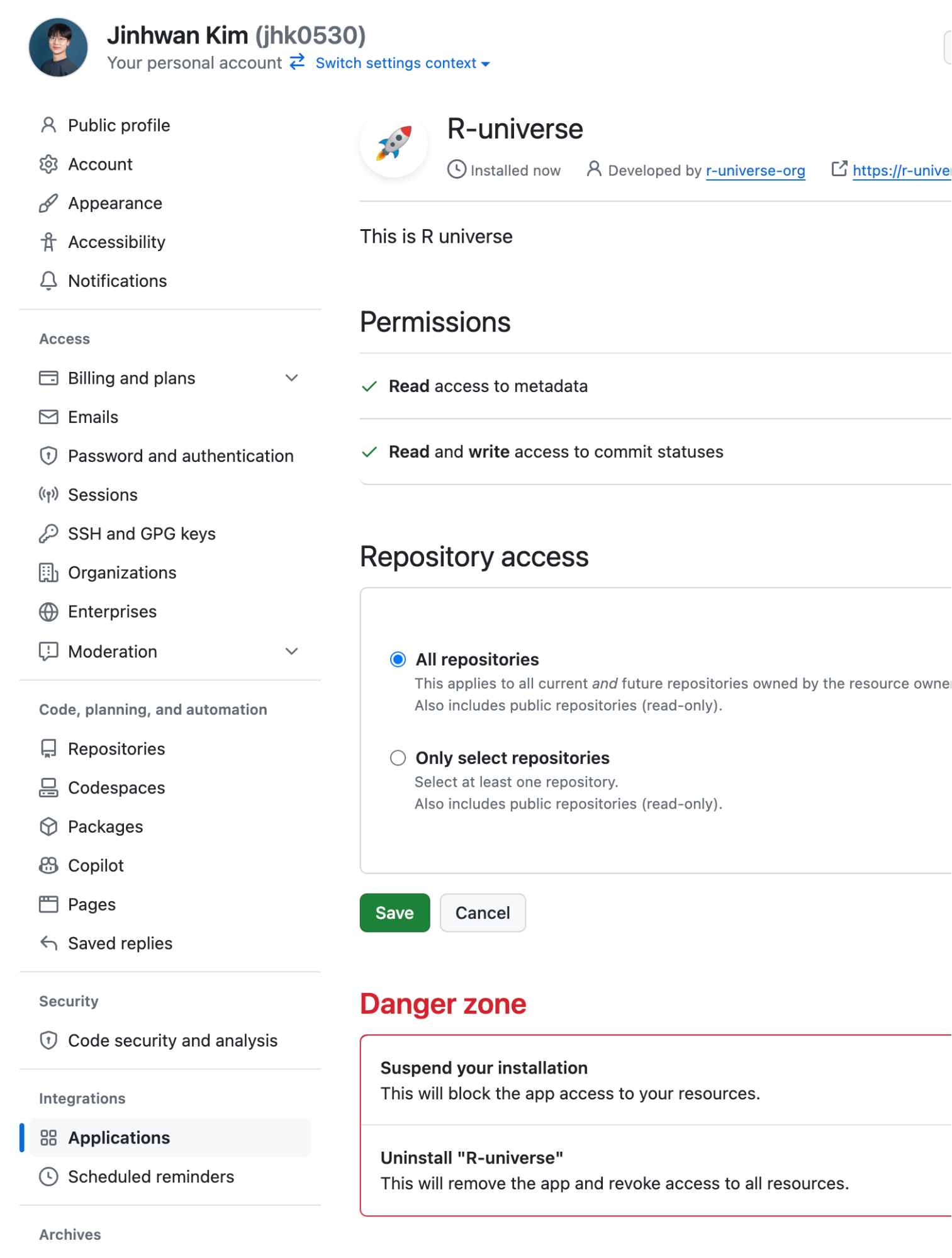
4) R-universe에 연동되는 동안 잠시 대기하기
깃허브 앱을 설치하고 나면 연동된 GHA들이 작동하며, 앞서 만든 jhk0530.r-universe.dev와 packages.json에 명시된 R 패키지 리포지토리를 훑고, 이를 r-universe/jhk0530 리포지토리로 동기화한다.
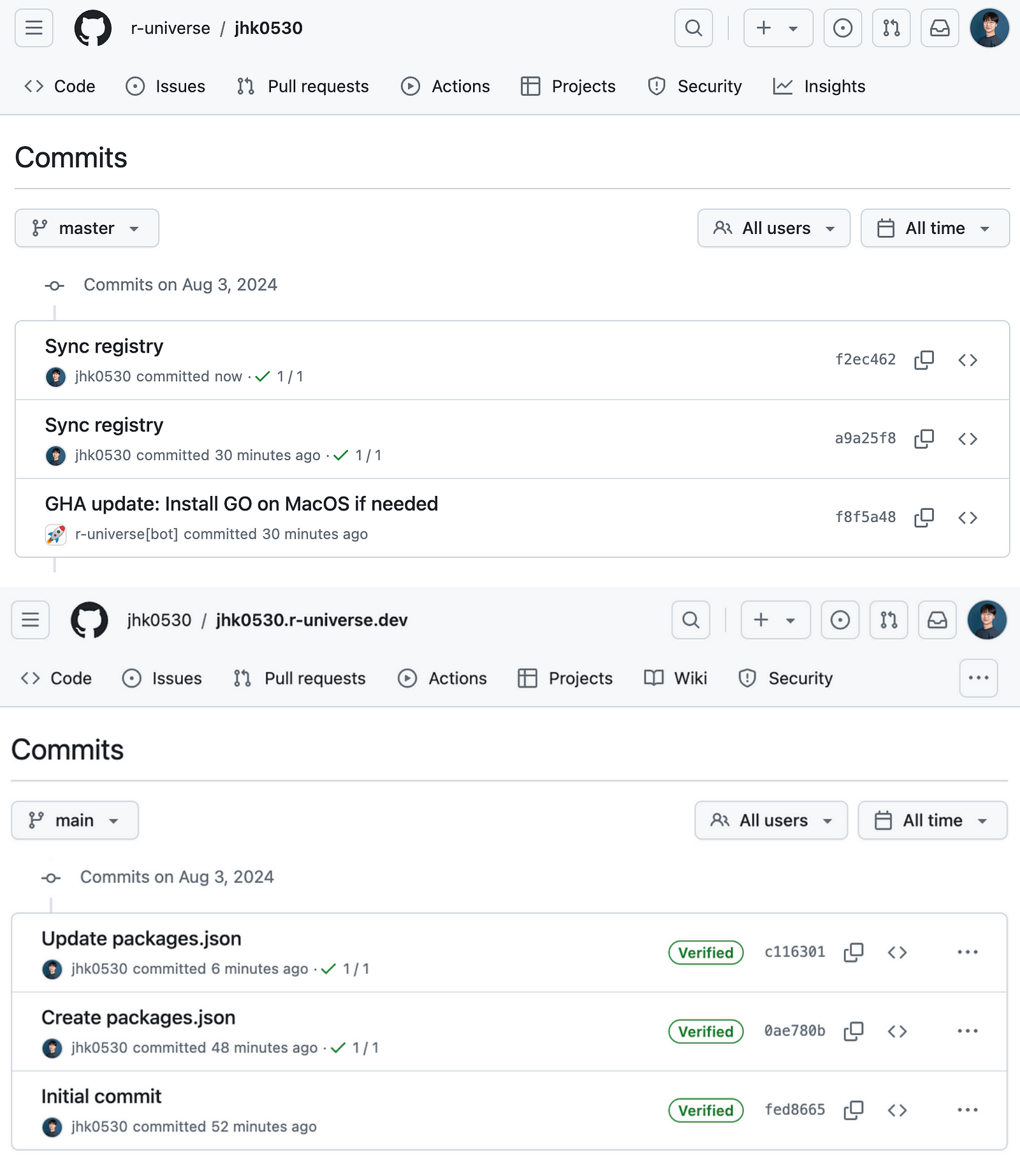
R 패키지 빌드를 포함한 GHA가 실행되며 약 30분 정도가 지난 후, jhk0530.r-universe/dev 페이지에서 내용이 업데이트된 것을 확인할 수 있다. 이때 packages.json에 적혀 있는 모든 R 패키지에 대해 GHA를 활용해 빌드 작업을 진행하므로, 만약 패키지 목록이 많다면 시간이 더 걸릴 수도 있다.
5) 결과 및 마무리
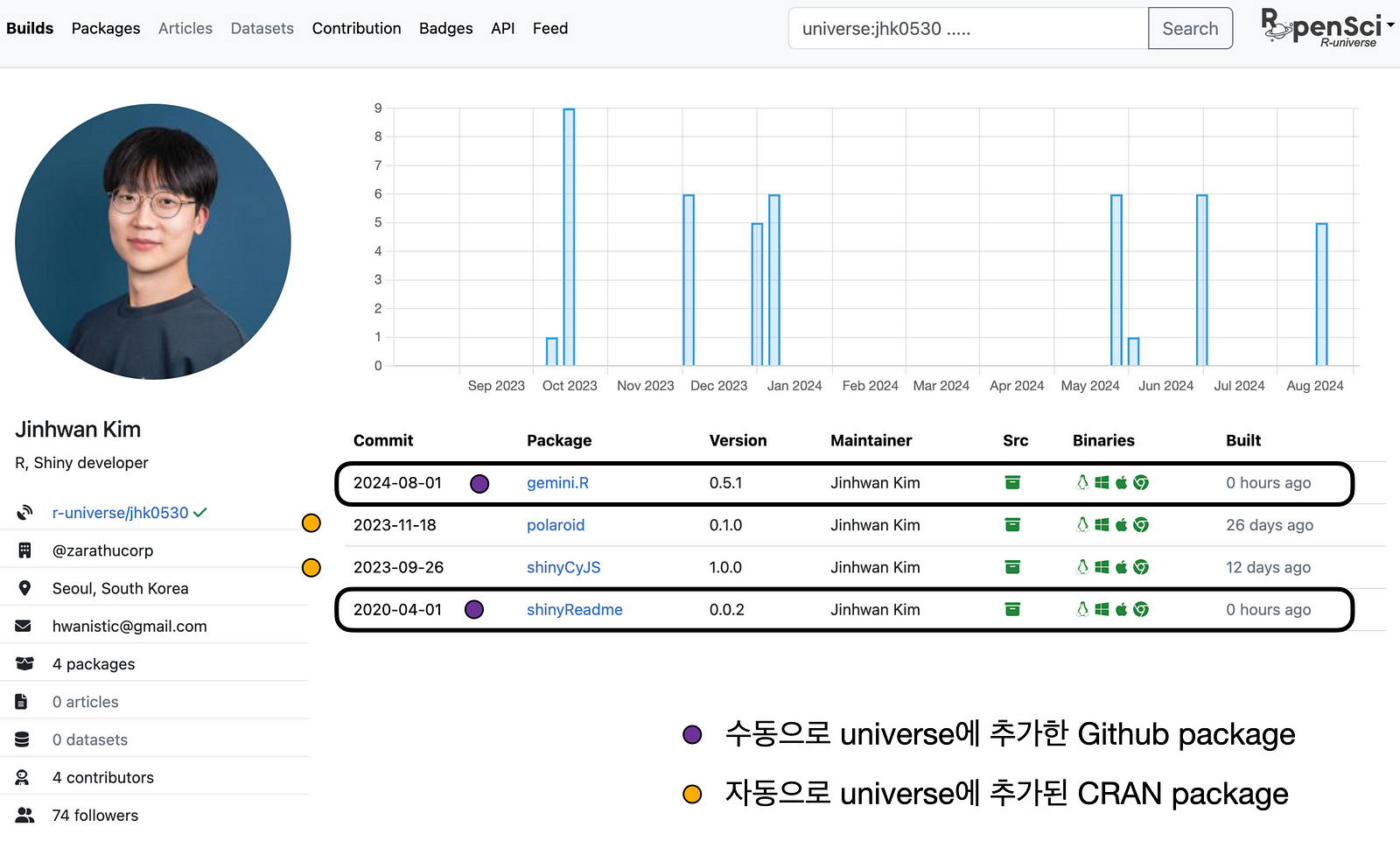
R-universe에는 미러로써 패키지의 빌드 상황을 보여주는 Builds 탭 외에도 Packages, 패키지에 다른 개발자와 기여를 주고받음을 보여주는 Contribution, 그리고 README.MD에 사용할 수 있는 배지(Badges) 이미지 등의 부가 기능이 있다.
이 중 Badges에 대해 간단히 소개해 보겠다.
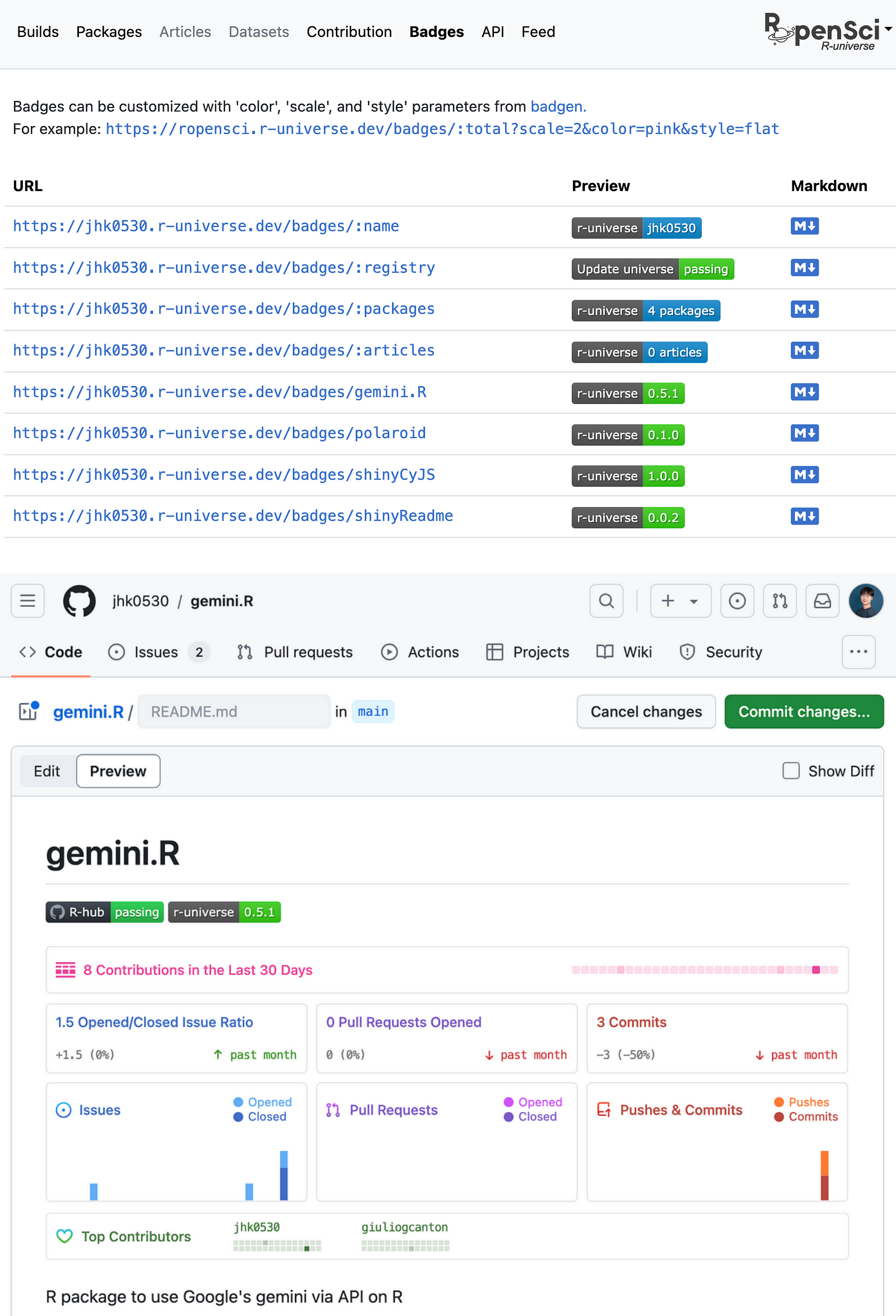
Badges에서는 내 universe의 상태, 그리고 각 패키지의 버전을 포함한 몇몇 지표를 복붙할 수 있는 코드로 보여준다. 이를 R 패키지의 README에 붙여주는 것으로 R-universe 업데이트 작업을 마무리할 수 있다.
이미지에 있는 또 다른 배지 R-hub는 여러 OS에서 R 패키지 빌드를 테스트할 수 있게 하는 기능으로, 이전에 쓴 글을 참고해 보면 좋다.
마치며
R-universe는 rOpenSci 재단에서 제공하는 개인 R 패키지 미러 역할을 하는 웹 서비스로, 만들기도 쉽고 비용이 들지도 않는다. 또한 CRAN뿐만 아니라 깃허브로 공개했던 비공식 R 패키지도 한꺼번에 관리할 수 있다.
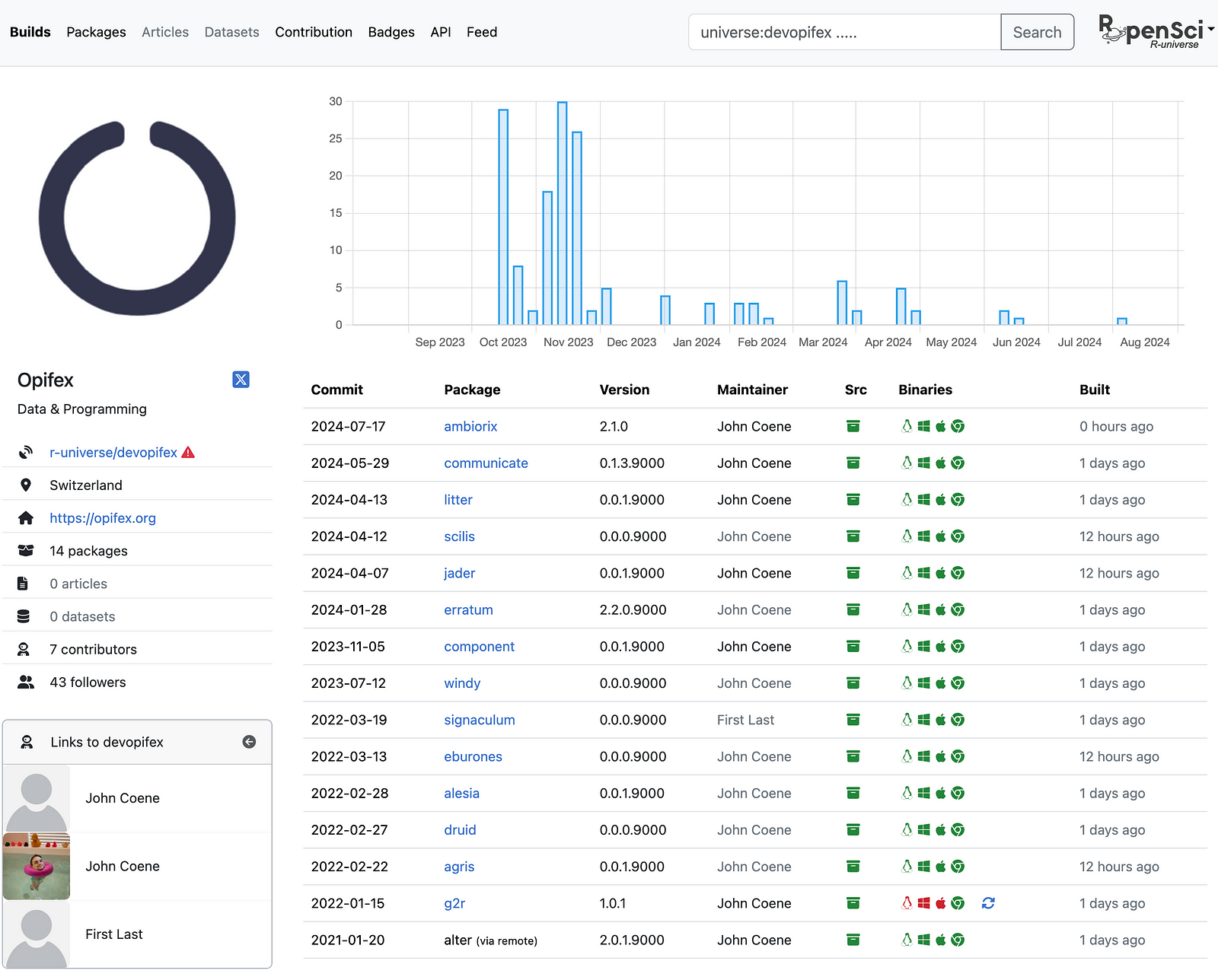
R-universe는 미러의 역할인 소스코드, OS별 빌드 결과, 패키지 다운로드, 더 편한 설치 외에도 여러 지표를 포함해 꽤 신기한 내용을 보여주기도 한다. 또한 사용자는 더 쉽고 편리하게 내 R 패키지를 찾거나, 사용할 수 있다. 개발자의 관점에서도 다른 R 개발자들이 어떻게 했는지를 보며 영감을 얻을 수 있다.
이에 더해 개인뿐만 아니라 Organization 레벨에서도 작업이 가능한 만큼, 최근 R, Shiny 개발자들이 각자의 universe 페이지를 부지런히 만들고 있다. 앞으로 ‘CRAN - 깃허브’ 또는 ‘CRAN - R-universe’처럼 제2의 방법으로 많이 활용될 것이다.